Bonjour à tous,
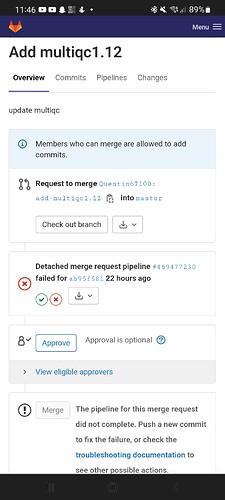
J'ai essayé d'ajouter multiqc 1.12 mais je me retrouve avec un "failed" sur gitlab.
A savoir qu'il y a quelques temps j'avais essayé d'ajouter seqkit, ça avait bugué donc j'avais laissé tombé et je suis passé par un conda perso, mais du coup la quand j'ai rajouté multiqc il a aussi créer le fichier méta pour seqkit.
Un peu d'aide ne serait pas de refus 
@team.software
Bonjour,
C'est sans doute parceque vos modifications pour les deux outils sont dans la meme branche git.
Comme indiqué dans la documentation: docs/beginner_guide.md · master · Quentin Chartreux / tools · GitLab
Il faut créer une nouvelle branche pour chaque nouvel ajout de package.
De plus il faut maintenir votre fork à jour, si vous souhaitez faire plusieurs proposition de changement étaler dans le temps.
Il me semble qu'il y'a des options de synchronisation dans la partie "settings" de gitlab. Sinon vous pouvez faire ca en ligne de commande, typiquement:
git remote add upstream https://gitlab.com/ifb-elixirfr/cluster/tools.git
git checkout master
git pull upstream master
git push -u origin master
Attention cela ne fonctionne que si vous n'avez fait aucune modification sur la branche master (c'est a dire quand vous créer une nouvelle branche pour chaque modification)
A noter aussi que si c'est trop compliqué, une solution plus simple consiste a supprimer le fork de votre compte pour ensuite le recréer. Il sera alors automatiquement à jour.
Bonjour,
Étant en vacances j'ai dû faire ça sur mon tél donc c'était plus simple de supprimer le projet et de refaire un fork.
En revanche pour la mise à jour automatique, il semble bien y avoir une option Ici cependant le sens pull n'est accessible que lorsqu'on a un gitlab payant donc je ne peux pas.
J'avais fait une deuxième merge request pour ajouter seqkit juste après multiqc mais du coup je suppose que je vais avoir le même problème, comme entre temps la MR de multiqc à été faite, la branche add-seqkit n'est plus à jour...
J'ai donc fermé la MR d'ajout de seqkit, et refait en utilisant la ligne de commande, c'est plus simple (surtout avec le script add_conda_tools qui créer les fichiers tout seul).
J'espère que cette fois ci j'ai tout fait comme il faut 
![]()