Bonjour,
Je reprends une analyse de données de RNAseq faite antérieurement par une collègue sous RStudio. L'idée est de transposer celle-ci en Jupyter Notebooks.
J'utilise le kernel R4.0.3 (le plus récent disponible sur IFB JupyterHub) mais il me manque certains packages :
- exprAnalysis
- PCAtools
- EnhancedVolcano
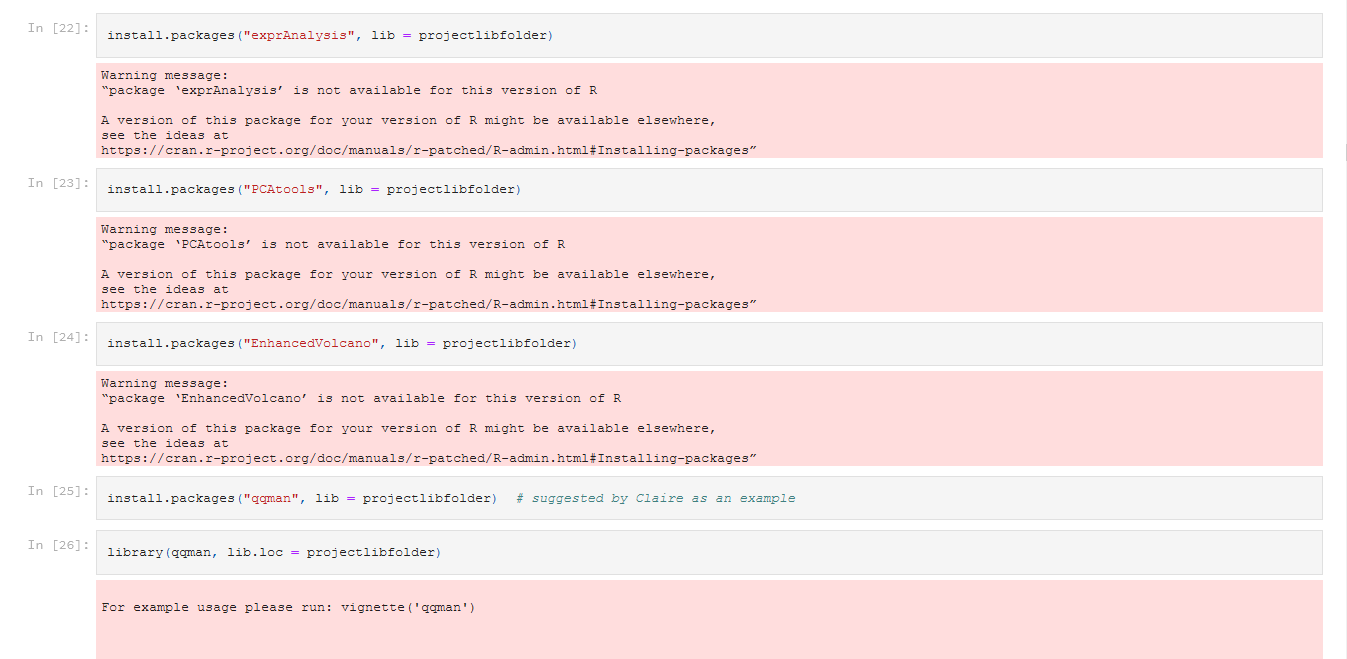
J'ai essayé de les installer dans un sous-dossier du projet (gonseq/Building/Results/Routputs/additionallibs) mais ils ne semblent pas être compatibles avec la version de R. Pourtant, le package "qqman" (pour test) s'installe correctement (cf pièce jointe
).
Merci pour votre aide et éclairage,
Pour information, je suis en congé demain jusqu'au 8/8 puis du 20/8 au 30/8.
Pas d'urgence 
Cordialement,
Bonjour Bénédicte,
Je suis pas certain de la bonne façon de faire (d'autres pourront sûrement me corriger ou compléter) mais une possibilité est d'utiliser Bioconductor ou install_github.
# https://github.com/ShirinG/exprAnalysis#installation
devtools::install_github("ShirinG/exprAnalysis", build_vignettes=TRUE, ref = "stable.version0.1.0")
# https://bioconductor.org/packages/release/bioc/html/PCAtools.html
BiocManager::install("PCAtools")
# https://bioconductor.org/packages/release/bioc/html/EnhancedVolcano.html
BiocManager::install("EnhancedVolcano")
# https://github.com/stephenturner/qqman#installation
devtools::install_github("stephenturner/qqman")
Par contre cela risque de générer des problèmes de dépendance qu'il va falloir résoudre petit à petit.
Pour les installer dans un sous-dossier du projet, je pense qu'il faut jouer avec les path (R_LIBS_USER peut-être) ou utiliser with_libpaths.
Bonnes vacances 
![]()