Bonjour
Plusieurs participantes du Diplôme Universitaire en Bioinformatique Intégrative ont rapporté des problèmes pour installer sur le cluster et/ou RStudio les packages de bioinformatique intégrative requis pour leur travail personnel:
Serait-il possible d'installer ces 3 packages d'une part sur la version R en ligne de commande du cluster, et d'autre part sur RStudio, pour éviter que chaque apprenant ne doive le faire (et apparemment échouer) ?
Merci
Jacques
Bonjour,
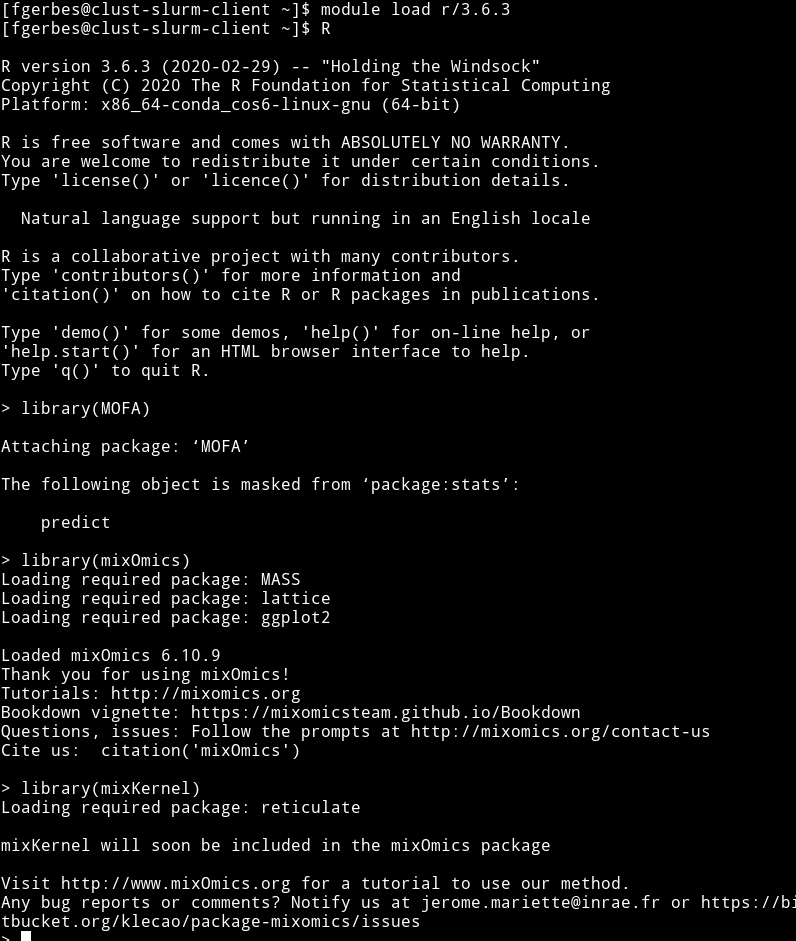
Normalement ces packages sont installés sur les versions r/3.5.1 et r/3.6.3 sur le cluster (et Rstudio utilise la r/3.6.3 du cluster, donc avec les mêmes packages)
Je viens de vérifier et ils se chargent bien:
Merci à @dbenaben qui a vérifié les versions:
r/3.6.3
packageVersion("MOFA")
[1] '1.2.0'
packageVersion("mixOmics")
[1] '6.10.9'
packageVersion("mixKernel")
[1] '0.4'
r/3.5.1
packageVersion("MOFA")
[1] '1.3.1'
packageVersion("mixOmics")
[1] '6.13.0'
packageVersion("mixKernel")
[1] '0.4'
Donc peut-être que le souci vient d'une version trop ancienne de ces packages sur r/3.6.3, on peux essayer de les mettre à jour.
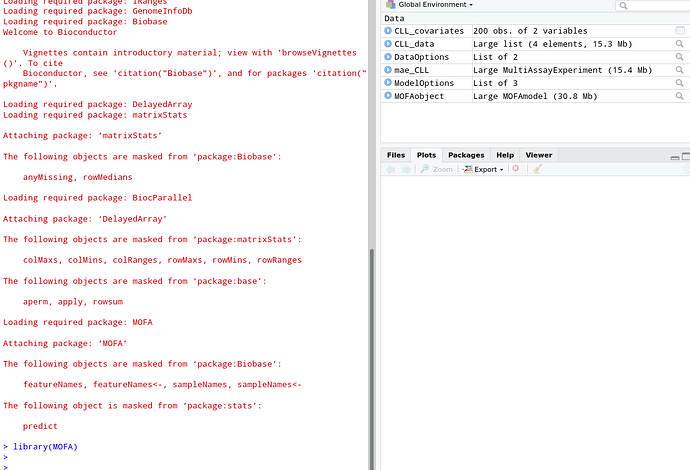
Apparement ca marche aussi dans Rstudio:
Pour MOFA:
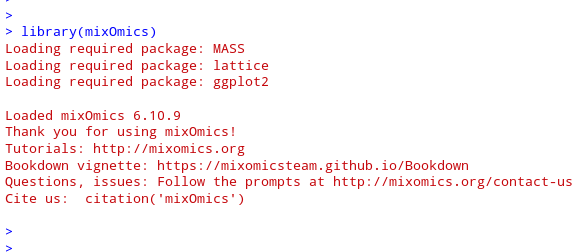
mixOmics:
mixKernel:

Merci pour le traitement rapide de la requête, @Francois et merci à @dbenaben pour l'installation.
Je vais demander aux participantes du DUBii de préciser la nature de leur problème, qu'elles avaient posté sur le slack de la formation.
PS: Je n'ai fait que vérifier les versions. C'est bien François et Gildas qui ont fait un gros travaille dessus