Bonjour,
Je vois que le logiciel Kraken est disponible en module load sur le cluster.
Est-ce qu'il y a une DB associée ou il faut se la créer soi-même ?
Merci d'avance,
Claire
Certaines DB précalculées sont téléchargeables depuis le github de kraken2 : https://benlangmead.github.io/aws-indexes/k2
Tu as combien d'identités @clairetn @ctn 
Est-ce que je peux me contenter de télécharger chaque archives et de les décompresser ?
Bonjour,
j'aurais aimé savoir si les bases de données avaient été téléchargées. Et si ces dernières étaient compatibles avec Kraken2 ? Car je ne pense pas que ça soit le cas...
Comme je dois utiliser Kraken2, je dois également utiliser une base de données spécifique au logiciel... Mais cette dernière est trop grande pour que je la télécharge provisoirement sur mon espace de projet ![]()
Si nécessaire voici les lignes de commande pour la base de données :
kraken2-build --download-library nt --threads 20 --db {chemin_de_stockage}/nt --use-ftp
kraken2-build --download-taxonomy --threads 20 --db {chemin_de_stockage}/nt --use-ftp
En vous remerciant par avance,
Aurélie
Jobs en cours

Merci beaucoup ! 
(re)Bonjour,
Je m'excuse de vous déranger mais j'aurais aimé savoir si la base de données avait été téléchargée sans problème.
Serait-il également possible de m'indiquer l'emplacement de cette dernière sur le serveur ? J'ai supposé qu'elle serait placée dans /shared/bank/nt/current mais ce n'est pas le cas... 
En vous remerciant d'avance,
Aurélie
Ah oui, désolé, j'ai oublié de vérifier.
Je crois que le job s'arrêté car il a passé les 24h 
Je relance sur la partition long
Pour suivre le job :
sacct -j 1491929
L'emplacement : /shared/bank/nt/nt_2021-01-29/kraken2
Merci !
Le job s'est terminé sans retourner d'erreurs. ![]()
Seulement la base de données ne semble toujours pas fonctionner... Lorsque je teste ma commande Kraken2, le logiciel me retourne l'erreur suivante :
kraken2: database ("/shared/bank/nt/nt_2021-01-29/kraken2/library/nt") does not contain necessary file taxo.k2d
Et j'ai beau rechercher dans les fichiers, je ne trouve pas ce fichier non plus.
Il semblerait que je me sois trompée dans l'ordre des commandes. Il faudrait apparemment faire [Lien] :
kraken2-build --download-taxonomy --db $DBNAME --use-ftp
puis
kraken2-build --download-library nt --db $DBNAME --use-ftp
kraken2-build --build --db $DBNAME
En m'excusant de mon erreur et en vous remerciant encore pour votre aide,
Aurélie
J'ai juste relancer la dernière étape vu que les 2 autres avait à priori tourné
Super merci !
Par contre il semblerait y avoir eu un soucis avec la mémoire vive ? ![]()
Failed attempt to allocate 243640340480bytes;
you may not have enough free memory to build this database.
Perhaps increasing the k-mer length, or reducing memory usage from
other programs could help you build this database?
build_db: unable to allocate hash table memory
xargs: cat: terminated by signal 13
sacct -j 15039003
Voyons si ça passe avec 500GB 
Sinon, on augmentera en effet la taille des k-mers
Croisons les doigts 




Désolée de vous embêter autant avec cette base de données !
$ ll /shared/bank/nt/nt_2021-01-29/kraken2
total 240866252
-rw-rw-r-- 1 glecorguille glecorguille 243640340512 Feb 21 17:08 hash.k2d
drwxrwxr-x 3 glecorguille glecorguille 4096 Feb 15 18:35 library
-rw-rw-r-- 1 glecorguille glecorguille 56 Feb 21 17:08 opts.k2d
-rw-rw-r-- 1 glecorguille glecorguille 2828369642 Feb 18 11:19 seqid2taxid.map
-rw-rw-r-- 1 glecorguille glecorguille 174637780 Feb 21 13:28 taxo.k2d
drwxrwxr-x 2 glecorguille glecorguille 4096 Feb 18 11:08 taxonomy
-rw-rw-r-- 1 glecorguille glecorguille 3537616 Feb 18 11:19 unmapped.txt
On a ce fameux taxo.k2d 
Bonjour,
J'ai essayé d'utilisé le mois passé la database associé au kraken disponible sur /shared/bank/nt/nt_2021-01-29/kraken2 mais cela ne marche pas.
Pourriez-vous m'aider svp?
Merci et bonne année2024.
BK.
Bonjour,
Si cela peut aider, voilà un code qui lance kraken2 avec des fq.gz paired-end (code relancé à l'instant pour vérifier que c'était toujours ok) :
#!/bin/bash
#SBATCH --partition long
#SBATCH --mem 230GB
DATADIR="/shared/projects/PATH_TO_YOUR_FASTQ_FILES/"
KRAKENDBDIR="/shared/bank/nt/nt_2021-01-29/kraken2/2021-02-02/"
SAMPLE_R1=${1}_R1.fq.gz
SAMPLE_R2=${1}_R2.fq.gz
SAMPLE=${1}
if [ -e ${DATADIR}${SAMPLE_R1} ]
then
module load kraken2
srun kraken2 --db ${KRAKENDBDIR} --report k2Report_${SAMPLE}.txt --use-names --output k2Output_${SAMPLE}.txt --gzip-compressed --paired ${DATADIR}${SAMPLE_R1} ${DATADIR}${SAMPLE_R2}
else
echo "error in input file: "${DATADIR}${SAMPLE_R1}
fi
à lancer avec sbatch kraken2_run.sh SRR12762560 (ici, les inputs *.fq.gz se nomment SRR12762560_R1.fq.gz et SRR12762560_R2.fq.gz)
Claire
Bonjour Claire,
Merci pour le script, il marche. Mais le fichier report est vide, est-ce à dire qu'il trouve pas de contaminant? Normalement il devrait me dire à quel type d'organisme correspond mon génome, est-ce cela?
Bonjour BK,
De mon côté, j'ai bien qqch dans mes fichiers kReport... :
| 14.52 | 1452 | 1452 | U | 0 | unclassified |
|---|---|---|---|---|---|
| 85.48 | 8548 | 1869 | R | 1 | root |
| 62.95 | 6295 | 69 | R1 | 131567 | cellular organisms |
| 51.33 | 5133 | 8 | D | 2759 | Eukaryota |
| ... | |||||
| 0.01 | 1 | 0 | R1 | 2787823 | unclassified entries |
| --- | --- | --- | --- | --- | --- |
| 0.01 | 1 | 0 | R2 | 12908 | unclassified sequences |
| 0.01 | 1 | 0 | R3 | 151659 | environmental samples |
| 0.01 | 1 | 1 | S | 155900 | uncultured organism |
ou kOutput...
Vu la ligne "unclassified entries" (nombreuses !) de la fin de mon fichier report, je suppose que ton report vide n'est pas du à l'absence de contaminant mais plutôt à un problème technique. As-tu regardé ce qu'il y a dans le fichier de sortie d'erreur (par défaut chez moi il s'appelle slrum-<numéroduJob>.out) ? Kraken2 y écrit qq lignes, il y aura peut-être une info expliquant ton fichier vide ?
Voici mon script
#!/bin/bash
#SBATCH --partition long
#SBATCH --mem=500G
DATADIR="/shared/ifbstor1/projects/b_pseudomallei/MG_Bpm_NewC/reads/2021/"
KRAKENDBDIR="/shared/bank/nt/nt_2021-01-29/kraken2/2021-02-02/"
SAMPLE_R1=${21_144_S1_L001}_R1_001.fq.gz
SAMPLE_R2=${21_144_S1_L001}_R2_001.fq.gz
SAMPLE=${21_144_S1_L001}
if [ -e ${DATADIR}${SAMPLE_R1} ]
then
module load kraken2
srun kraken2 --db ${KRAKENDBDIR} --report k2Report_${SAMPLE}.txt --use-names --output k2Output_${SAMPLE}.txt --gzip-compressed --paired ${DATADIR}${SAMPLE_R1} ${DATADIR}${SAMPLE_R2}
else
echo "error in input file: "${DATADIR}${SAMPLE_R1}
fi
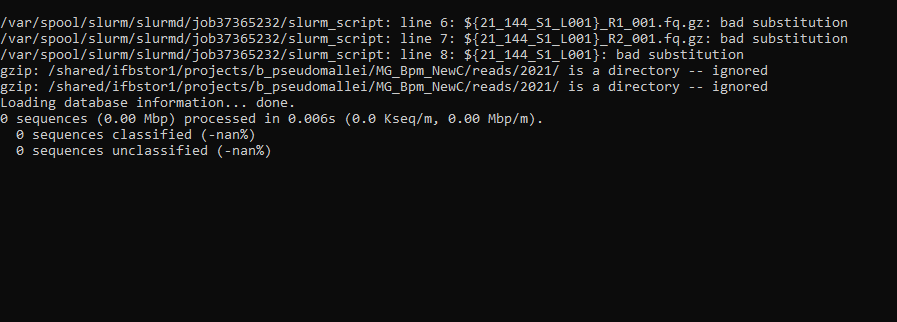
Et voilà ce qui est écris dans le fichier erreur
/var/spool/slurm/slurmd/job37365232/slurm_script: line 6: ${21_144_S1_L001}_R1_001.fq.gz: bad substitution
/var/spool/slurm/slurmd/job37365232/slurm_script: line 7: ${21_144_S1_L001}_R2_001.fq.gz: bad substitution
/var/spool/slurm/slurmd/job37365232/slurm_script: line 8: ${21_144_S1_L001}: bad substitution
gzip: /shared/ifbstor1/projects/b_pseudomallei/MG_Bpm_NewC/reads/2021/ is a directory -- ignored
gzip: /shared/ifbstor1/projects/b_pseudomallei/MG_Bpm_NewC/reads/2021/ is a directory -- ignored
Loading database information... done.
0 sequences (0.00 Mbp) processed in 0.006s (0.0 Kseq/m, 0.00 Mbp/m).
0 sequences classified (-nan%)
0 sequences unclassified (-nan%)
Merci pour ton aide.
BK.